Bootstrapping: Unterschied zwischen den Versionen
Wehner (Diskussion | Beiträge) Keine Bearbeitungszusammenfassung |
Wehner (Diskussion | Beiträge) Keine Bearbeitungszusammenfassung |
||
| Zeile 27: | Zeile 27: | ||
Manly, B.F. (2018). ''Randomization, bootstrap and Monte Carlo Methods in biology''. Chapman and Hall/CRC | Manly, B.F. (2018). ''Randomization, bootstrap and Monte Carlo Methods in biology''. Chapman and Hall/CRC | ||
Rudolf, M., & Kuhlisch, W. (2008). ''Biostatistik: Eine Einführung für Biowissenschaftler'' (Kapitel | Rudolf, M., & Kuhlisch, W. (2008). ''Biostatistik: Eine Einführung für Biowissenschaftler'' (Kapitel 5.5). München: Pearson Studium. | ||
Version vom 5. März 2020, 23:04 Uhr
Die Bootstrap-Technik nach Efron (1979) ist ein Resampling-Verfahren, welches es ermög-licht, Parameter aus einer Stichprobe zu schätzen, ohne Annahmen über eine spezifische Verteilung zu treffen. Sie wird in der Praxis angewandt, wenn die untersuchten Werte keiner bekannten Verteilung folgen.
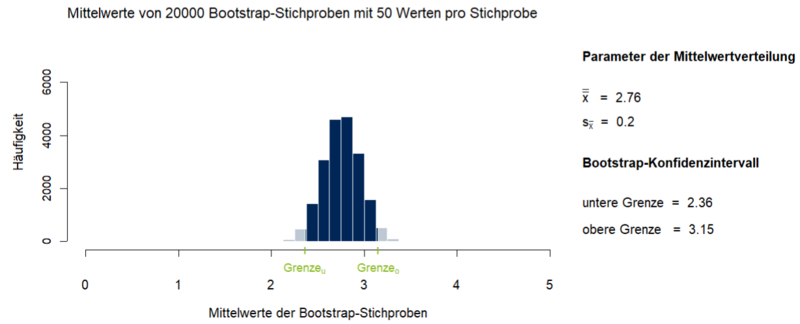
Das Prinzip des Bootstrapping beruht darauf, Informationen über eine unbekannte Po-pulation durch Resampling einer einzelnen Stichprobe aus dieser Population zu erhalten. Resampling bedeutet in diesem Fall, dass eine große Anzahl Bootstrap-Stichproben mit Zu-rücklegen aus der Ausgangsstichprobe gezogen werden. Dabei wird zunächst ein einzelner Wert gezogen und wieder in die Ausgangsstichprobe zurückgelegt. Dieser Vorgang wird so häufig wiederholt, bis die Bootstrap-Stichprobe die Größe der Ausgangsstichprobe erreicht hat. Durch das Zurücklegen kann eine Bootstrap-Stichprobe einzelne Werte der Ausgangs-stichprobe mehrfach, andere Werte wiederum gar nicht enthalten. Auf diese Art werden übli-cherweise mehrere tausend Bootstrap-Stichproben erzeugt. Für jede dieser Bootstrap-Stichproben kann z.B. der arithmetische Mittelwert x̅ oder ein anderer interessierender Para-meter geschätzt werden. Anhand der Verteilung der arithmetischen Mittelwerte der Bootstrap-Strichproben sind Rückschlüsse auf die Parameter der Population möglich. Eine häufige An-wendung ist die Berechnung valider Konfidenzintervalle für Parameter der Population. In Ab-bildung 1 sind die Mittelwerte und das 95%-Konfidenzintervall von 20000 Bootstrap-Stichproben dargestellt, die aus einer nicht-normalverteilten Ausgangsstichprobe mit einem Mittelwert von x̅ = 2.75 und einer Standardabweichung von s = 1.45 gezogen wurden. 95 % der Mittelwerte aller gezogenen Bootstrapstichproben befinden sich zwischen 2.36 und 3.15.
Als non-parametrisches Verfahren ohne Verteilungsannahmen ist Bootstrapping in vielen Kon-texten anwendbar, wie z.B. bei der Prüfung von indirekten Effekten bei linearen Strukturglei-chungsmodellen. Eine wichtige Voraussetzung für die Anwendung der Bootstrap-Technik ist jedoch, dass die Ausgangsstichprobe repräsentativ für die Grundgesamtheit sein muss. Au-ßerdem ist das Verfahren rechenintensiv, was besonders bei zunehmender Anzahl an Bootstrap-Stichproben berücksichtigt werden muss.
kkk Im Video wird die Bootstrap-Technik näher erläutert.
kkk Die Methode des Bootstrappings lässt sich in der interaktiven Simulation mit verschiedenen Ausgangsstichproben und für verschiedene Stichprobengrößen und Konfidenzniveaus nachvollziehen.
Weiterführende Literatur
Manly, B.F. (2018). Randomization, bootstrap and Monte Carlo Methods in biology. Chapman and Hall/CRC
Rudolf, M., & Kuhlisch, W. (2008). Biostatistik: Eine Einführung für Biowissenschaftler (Kapitel 5.5). München: Pearson Studium.